6篇Nature齐发!癌症研究和云计算的里程碑
撰文 | BioArt编辑部
编者按:除了Nature一次性发表6篇论文外,Nature Genetics杂志发表了3篇,Nature Communications发表9篇,Nature Biotechnology一篇,详细报道BioArt后续会陆续发布。文章合集网址:https://www.nature.com/collections/afdejfafdb
图片来源:https://www.nature.com/articles/d41586-020-00308-w
2001年,人类基因组计划完成,整个计划花费约30亿美元,1985年提出,1990年正式启动,历经十余年。肿瘤是最热门的研究领域之一,该领域的研究者们一直希望可以借助测序手段去表征肿瘤的基因组,从而一窥肿瘤的秘密。在随后的20年中,各种测序技术的发展以及测序成本的降低使得实现上述目标变的可能。
2006年,美国国家癌症研究所和国家人类基因组研究所开启了癌症基因组图谱计划(The Cancer Genome Atlas Program),简称TCGA。该计划涵盖了33种癌症类型,包括超过2万个癌症及正常组织的分子特征。经过12年的发展,TCGA产生了超过2.5PB的海量数据,包括基因组、表观组、转录组和蛋白质组。

随着TCGA的发展以及各种关于癌症研究的深入,大家越来越认识到详细记录癌症的每一个突变是可行的。于是在2008年,全球癌症基因组协会(global cancer genomics community)建立了International Cancer Genome Consortium(国际癌症基因组联盟,ICGC),ICGC的目标是系统性记录各种常见癌症中的突变(ICGC成立初期计划投资10亿美元,用10年时间详尽、深入研究可导致癌症的变异基因,集世界各国和地区的科学家之力,绘制完整的人类癌症基因图谱,是一项与人类基因组计划具有同等重要意义的研究计划——引自http://www.cncbd.org.cn/News/Detail/1261)。在人员上,ICGC与TCGA有着一定的重叠。每一个ICGC小项目瞄准一个癌症或者相似的一类癌症。
TCGA与ICGC的早期研究揭示,在不同肿瘤中,突变似乎存在有一定的共性和差异,比如不同肿瘤中都有同一个突变,有些突变只在特定的一个或几个肿瘤中存在,这些发现揭示了肿瘤背后是复杂的,再加上不同的小组在处理各自的项目中存在有各种各样的技术障碍甚至有些标准参差不齐,因此为达到泛癌分析以及实现标准化,Pan-Cancer Analysis of Whole Genomes(PCAWG) Consortium应运而生。
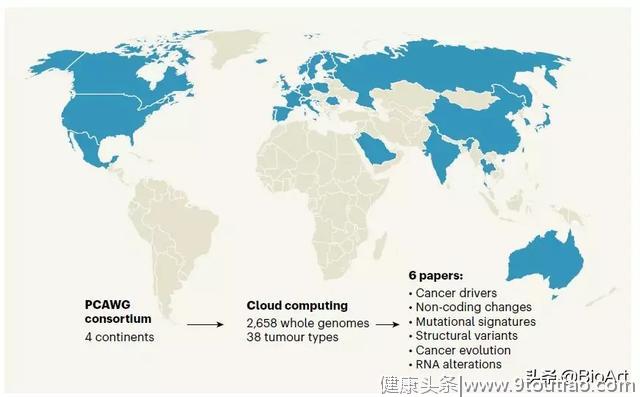
PCAWG联盟由4大洲744个机构的科研人员所组成,分为16个小组,每个小组专注于肿瘤基因的不同方面。该联盟需要克服整个项目所面临的技术、伦理、道德、法律等方面的障碍;在工作内容上,主要整合分析38种肿瘤类型,获得了2658个肿瘤全基因组。这一工作涉及到海量的数据处理,需要多种算法相的结合以及多数据中心的联合。当然,这一项目也证明了国际之间在云计算方面合作的可能性。
2020年2月4日,全基因组泛癌分析(PCAWG)联盟在Nature杂志上连发6篇文章,提出了目前为止最为全面的癌症基因组分析。与以往关注于蛋白编码区不同,这次是分析癌症全基因组。
Nature杂志对6篇系列工作给予高度重视,发表了相关社论
该项目最后以6篇Nature文章呈现出来,涵盖肿瘤驱动突变、非编码区域、突变特征、结构变异、肿瘤进化和RNA改变共计六个方面。下面将简单概括这六篇文章,同时在后续报道中我们会进一步详细介绍这些内容。
一、驱动突变
第一篇文章来自于The ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium,标题为Pan-cancer analysis of whole genomes。该文章从整体上描述了38种肿瘤类型2658个肿瘤全基因组,展示了PCAWG项目相关数据的广度与深度。据分析,平均每一个癌症基因组携带有4-5个驱动突变,然而大约5%没有发现,从另一方面讲有可能目前对于驱动突变的了解或者发现并不完全;另外许多肿瘤中表现出chromoplexy (17.8%)和 chromothripsis (22.3%),而这会导致基因组结构的改变。

二、非编码区域改变
第二篇文章来自于PCAWG Consortium和Joachim Weischenfeldt, Rameen Beroukhim,Iñigo Martincorena, Jakob Skou Pedersen, Gad Getz,题目为Analyses of non-coding somatic drivers in 2,658 cancer whole genomes。该文章分析了2658个肿瘤基因组中非编码区域的驱动突变和结构变异。鉴于非编码区域的复杂性,研究人员开发了不同的算法系统性鉴别上述变异,确定了以往报道的驱动突变,也怀疑了以往部分突变,并发现了一些新的驱动突变。比如TP53基因非编码区的一个复发突变,TRET非编码区的一个突变与该酶过表达有关等。虽然这些非编码区的突变频率不如编码区,但是这些突变也至关重要。

三、突变特征
第三篇文章来自于PCAWG Consortium和Steven G. Rozen, Michael R. Stratton,题目为The repertoire of mutational signatures in human cancer。体细胞突变可由多种原因造成,包括外源因素和内源因素。那么这些突变特征是什么呢?研究者从4645个全基因组测序和19184个外显子测序获得的84,729,690个体细胞突变中分析得到了81个突变特征:49个单碱基替换特征、11个双碱基替换特征、4个群碱基替换特征和17小插入/缺失特征。这些突变特征既有以往报道的,也有这次新发现的。通过对这些突变特征的深入分析,挖掘出了不同突变特征与不同分子事件的关系,尽管许多突变特征没有明显诱因,但是这一系统性分析有利于我们了解肿瘤的发育与进化。

四、结构变异
第四篇文章来自于PCAWG Consortium和Joachim Weischenfeldt, Rameen Beroukhim,Peter J. Campbell,题目为Patterns of somatic structural variation in human cancer genomes。上一篇文章关注了以somatic mutation为内容的突变特征,在这篇文章中,研究者们关注了结构变异,发现了16个结构变异特征,比如缺失大小差异,在不同肿瘤中也有不同,在晚期复制区域富集;串联复制也有大小差异,在早期复制区域富集。

五、肿瘤进化
第五篇文章来自于PCAWG Consortium和Peter Van Loo, Moritz Gerstung, 题目为The evolutionary history of 2,658 cancers。在该项研究中,研究人员利用数据库中的全基因组测序数据,重建了突变进程和驱动突变的进化历史,发现早期肿瘤发生与少数驱动突变和拷贝数变异有关,但在随后肿瘤的发展过程中,突变图谱的变化则很大,在晚期阶段驱动基因突变的多样性可以达到4倍以上,同时基因组的不稳定性也增加。这一研究有助于了解肿瘤进化和帮助肿瘤早期诊断。

六、RNA改变
第六篇文章来自于PCAWG Consortium和Alvis Brazma, Angela N. Brooks,Gunnar Rätsch, 题目为Genomic basis for RNA alterations in cancer。RNA改变包括:过表达、异常剪接、RNA融合等。通过结合全基因组数据和转录组数据,研究人员发现了649个影响基因表达的单核苷酸突变,1900个与突变相关的异常剪接,82%的基因融合与结构变异有关。这些转录改变在不同肿瘤类型中有不同的特征,同时这些数据与基因组数据结合可以更好地让我们了解癌症中基因的功能。

观点与评论
在同期,密歇根大学的Marcin Cieslik与Arul M. Chinnaiyan发表了评论文章Global cancer genomics project comes to fruition对这一项目进行了简单的描述与总结。

这六篇文章与相关的文章是癌症研究和云计算的一个里程碑。这些研究无疑扩展了我们对癌症测序数据的理解,丰富了我们对癌症发生发展的认识,但是需要注意的是,这些研究也有一定的不确定性。另外,这些研究的一个局限是:缺少临床信息的支撑,比如治疗方法、临床收益等。在另外一个项目——International Cancer Genome Consortium–Accelerate Research in Genomic Oncology (ICGC–ARGO)中,则以超过10万个肿瘤患者为对象,加入了上述临床信息。我们也期待着这一项目能够更加丰富我们对于癌症的认识。
六篇Nature原文链接:
https://www.nature.com/collections/afdejfafdb
https://doi.org/10.1038/s41586-020-1969-6
https://doi.org/10.1038/s41586-020-1965-x
https://doi.org/10.1038/s41586-020-1943-3
https://doi.org/10.1038/s41586-019-1913-9
https://doi.org/10.1038/s41586-019-1907-7
https://doi.org/10.1038/s41586-020-1970-0
"