癌症背后谁在操控细胞功能?

世界上没有两片完全相同的树叶,也没有两个完全相同的癌症。基因测序研究表明,肺癌患者平均每人突变数目接近5000 个,且每个人突变的组合都不同,每个患者的基因组都是特异的。中国有60 万肺癌患者,其实更像60 万种不同的疾病。
在人人谈“癌”色变的时代,由于对其发病机理的科学认知有限,严重阻碍了肿瘤疾病的预判和诊治。然而自精准医疗提出后,基因测序和数据分析技术的进步为肿瘤致病机理的深入研究提供了新的思路。越来越多技术手段被应用到癌症研究中,多项研究揭示三维基因组结构在肿瘤细胞的基因表达和功能调控中的重要作用。关于癌症背后谁在操控细胞功能调控的疑问,基因组的三维结构研究让我们又有了新的发现。


基因组的三维结构与癌症
大量研究表明,癌症的发生发展与三维基因组结构异常有关,基因的缺失、易位、DNA拷贝的获得和丢失都会让染色体非常的混乱[1]。在我们已经了解基因组序列在癌症中是如何突变的信息后,研究热点开始逐步转向关注三维基因组如何参与癌症的发生和发展。
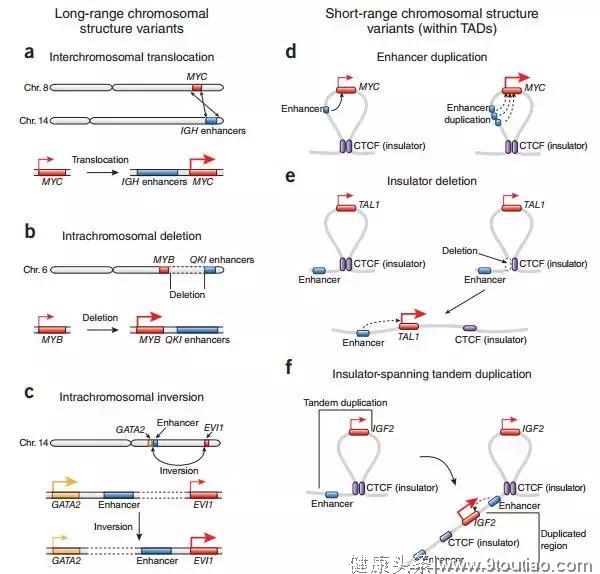
图1 非编码区域拷贝数异常导致致癌基因的过度表达
作为三维基因组学研究的利器,Hi-C测序技术近年来被广泛应用于肿瘤三维结构的研究,小编今天汇总了Hi-C技术在肿瘤三维基因组研究中的案例分享给大家,希望能为肿瘤研究的小伙伴们提供参考~

癌症中三维基因组的异常结构
1 | 多发性骨髓瘤[2]
样本:骨髓瘤细胞系RPMI-8226和U266,正常B细胞GM12878
多组学关联:Hi-C、WGS、RNA-seq、ChIP-seq
主要结论:
- 癌症三维基因组需要校正拷贝数的影响。
- 癌症基因组上存在大量染色体之间的相互作用的热点,这些热点反映了两个位点发生了易位。
- 拷贝数变异发生的边界与TAD的边界趋向重合。
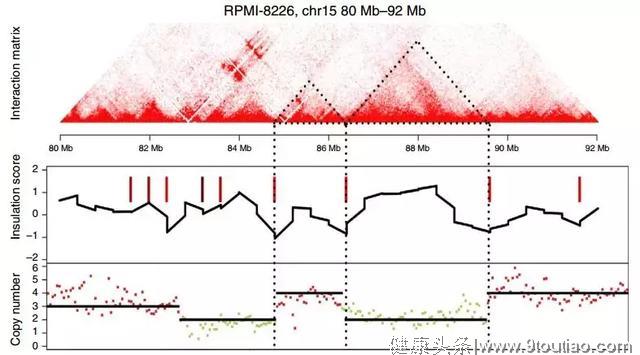
图2 拷贝数变异断裂位点与TAD边界容易重合
2 | 睾丸生殖细胞瘤[3]
样本:睾丸生殖细胞瘤(TGCT)细胞系NTERA2
多组学关联:Hi-C、3C、GWAS、RNA-seq、ChIP-seq
主要结论:
- 通过GWAS与已发表SNPs数据确认19个新的TGCTs 风险SNPs,将与睾丸癌相关的基因变化标志(包括SNPs和候选因果基因)增至44个。
- 许多基因变异通过干扰细胞内基因活化制造睾丸癌发病风险,那些容易受影响的基因可能参与了染色体稳定,或是与KIT癌基因(KIT–MAPK信号通路)有交互作用。
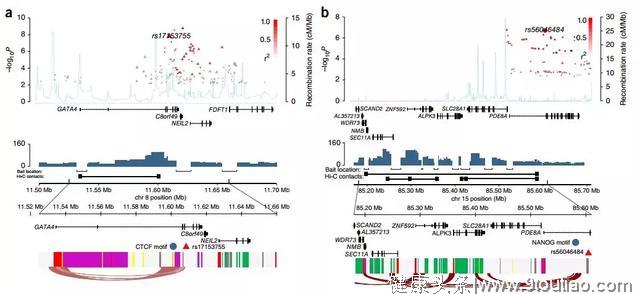
图3 风险位点的Hi-C loop分析
3 | 结直肠癌[4]
样本:结直肠癌(CRC)细胞系 LS174T、LoVo、Colo205
多组学关联: Capture Hi-C、RNA-seq
主要结论:
通过捕获Hi-C技术,对14个结直肠癌风险位点进行互作分析,鉴定这些风险位点与重要的远距离互作元件间的顺式和反式相互作用。

图4 捕获区域内的相互作用热图
4 | 乳腺癌[5]
样本:乳腺癌细胞系MCF-7和乳腺上皮细胞系MCF-10A
多组学关联:Hi-C、RNA-seq
主要结论:
- 小染色体中(16-22号染色体)MCF-10A细胞系中有较强的基因相互作用。
- MCF-10A、MCF-7染色体中部分A/B compartment结构发生传化,这些转化区域中的很多基因和癌症重要通路WNT相关。
- 鉴定TAD边界,发现乳腺癌细胞系特有的TAD边界。
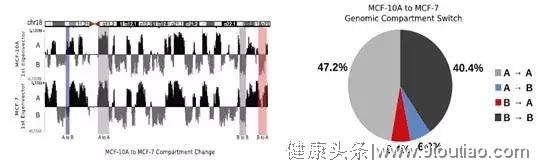
图5 A/B compartment 差异分析
5 | 前列腺癌[6]
样本:正常前列腺上皮细胞PrEC和2个前列腺癌细胞系PC3、LNCap
多组学关联:Hi-C、RNA-seq、ChIP-seq
主要结论:
- 前列腺癌细胞中,TADs 更小,数量增多。
- 包含TP53基因座的片段的缺失促进形成了新的TAD结构。
- 癌细胞特有的TAD边界富含CTCF和H3K4me3。
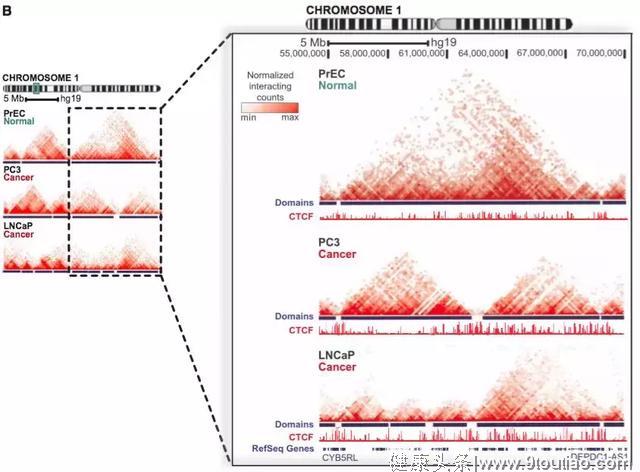
图6 健康细胞系和癌细胞系染色体互作热图

肿瘤Hi-C研究思路
为了方便大家参考,整理了一套肿瘤Hi-C研究思思路。
1. 材料选择:疾病细胞/组织、正常细胞/组织
2. 测序策略:建议分辨率5-40kb,Illumina PE150测序
3. 多组学策略:RAN-seq、ChIP-seq、WGS、ATAC-seq
4. 研究思路:
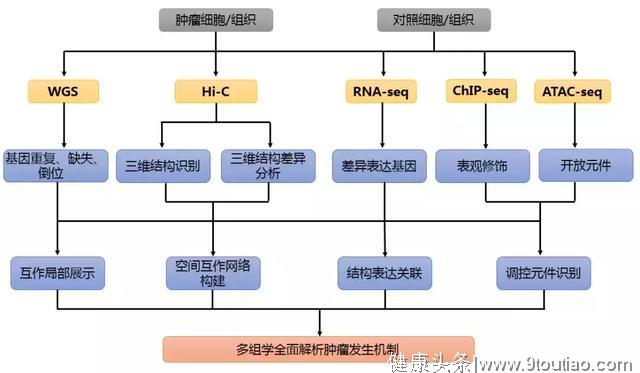
图7 肿瘤Hi-C多组学研究策略
人类基因组的 DNA 序列中蕴藏着远超出我们所想象的信息,三维基因组技术极大地促进了对癌症和复杂疾病的研究,但我们对人类三维基因组结构的认识才刚刚起步,更多科学的宝藏正等待科研工作者们去挖掘。Hi-C研究一小步,精准医疗一大步~
参考文献
[1] Beroukhim R, Zhang X, Meyerson M, et al. Copy number alterations unmasked as enhancer hijackers[J]. Nature Genetics, 2016, 49 (1) :5
[2] Wu P, Li T, Li R, et al. 3D genome of multiple myeloma reveals spatial genome disorganization associated with copynumber variations[J]. Nature Communications, 2017, 8 (1).
[3] K Litchfield, M Levy, G Orlando, et al. Identification of 19 new risk loci and potential regulatory mechanisms influencing susceptibility to testicular germcell tumor[J]. Nature Genetics, 2017, 49 (7) :1133-1140
[4] Mifsud B, Tavares-Cadete F, Young A N, et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C[J]. Nature Genetics, 2015, 47.
[5] Barutcu AR, Lajoie BR, Mccord RP, et al. Chromatin interaction analysis reveals changes in small chromosome and telomere clustering between epithelial and breast cancer cells[J]. Genome Biology, 2015, 16 (1) :1-14
[6] 6.Phillippa C. T, Joanna AK, Aaron T.L. L, et al. Three-dimensional disorganization of the cancer genome occurs coincident with long-range genetic and epigenetic alterations[J]. Genome Biology, 2015.26:719–731.