【Nature子刊】大数据精细定位,确定191个乳腺癌的靶基因!
导 读:乳腺癌是危害女性健康的头号恶性肿瘤,其发病率高、病因不明、早期症状不清。2017年,多项研究通过已全基因组关联分析(GWAS)寻找到150个乳腺癌相关的基因易感区段,但由于随机采样导致的抽样误差以及单核酸多态分子(SNP)之间复杂的连锁不平衡,GWAS定位到通常只是致病位点的相关区域,其结果具有一定复杂性和不准确性。
基于以上研究,剑桥大学的Alison Dunning课题组1月8日于Nature Genetics发表工作,分析了乳腺癌组织联合会(BCAC)中217,000多名受试者的基因组数据,通过精细定位技术(fine-mapping)在原先发现的150个易感基因区段中准确定义了191个风险基因,为临床癌症风险评估提供可靠依据,同时为乳腺癌发生机制的研究指明新的方向!

首先,研究者对样本进行基因分型,并使用多项式逻辑回归来识别每个易感区段中独立的乳腺癌关联信号,在每个信号中定义可信的因果变异(CCV)。通过联合关联分析与计算机基因组特征注释,结合贝叶斯方法和PAINTOR,研究者定义了205个具有可信因果变体的独立风险相关信号,并依据基因表达,染色质相互作用和功能注释将其进行排序。
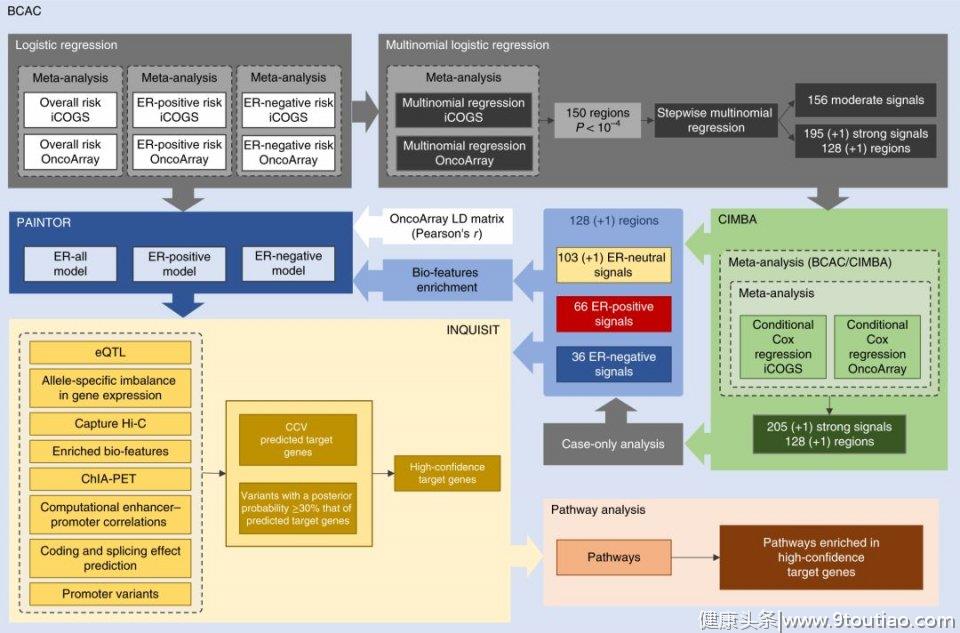
研究流程总结图
在确定每个信号中潜在的因果变异后,研究者希望揭示其靶基因以及在DNA水平上的功能。其中,研究者发现与雌激素受体(ER)阳性乳腺癌的特征变异,标记在ER阳性细胞上的开放活跃的基因调控区域,很大一部分与转录因子蛋白和调节因子的结合位点重叠。
另外,研究者还发现9种与ER阳性乳腺癌相关的重要靶基因编码蛋白质,这些蛋白产物在雌激素信号传导中发挥重要作用:CREBBP, EP300, ESR1, FOXI1, GATA3, MEF2B, MYC, NRIP1 和TCF7L2,其中CREBBP,EP300,ESR1,GATA3和MYC已被证明是癌症的驱动基因。
与ER阳性信号相反,研究者发现的ER阴性信号的基因组特征较少(这受限于样本数较少和可用的ChIP-Seq数据),但这同时也提示两种乳腺癌的发展具有共同分子机制。据此,研究者确定了35个ER阴性乳腺癌的靶基因,其中,CASP8和MDM4已有相关的功能研究。
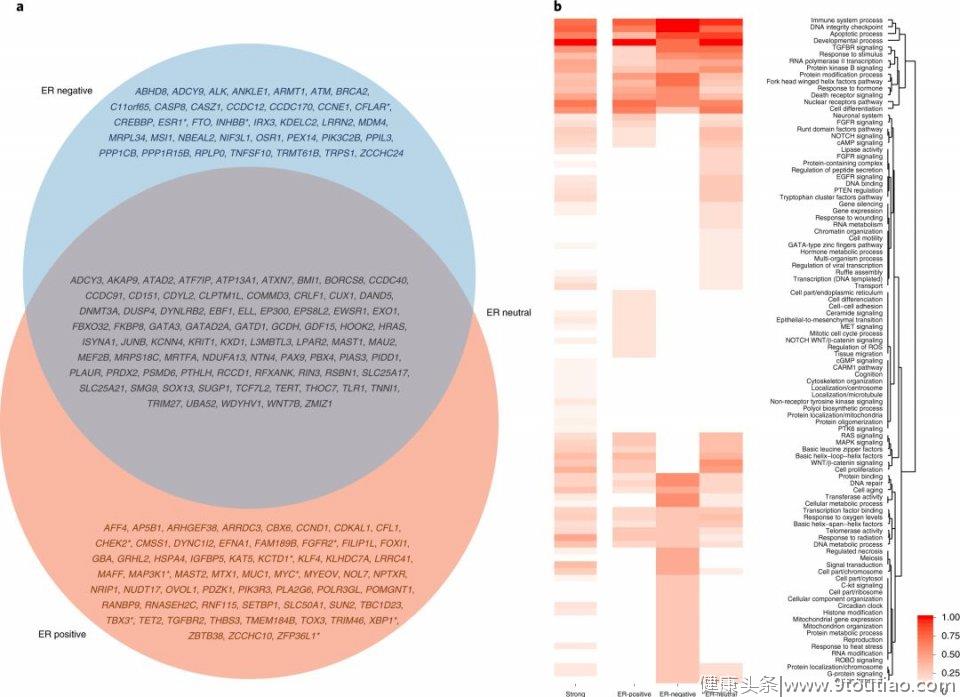
通过表型和显著通路预测靶基因
最后,研究者们通过分析基因的分子功能、细胞组分和参与的生物学过程这样一个“基因本体论(GO)”途径来发现新的靶基因。值得注意的是,14%的靶基因与免疫系统途径有关,在ER阴性的乳腺癌相关基因中这个比例达到19%,尤其是涉及T细胞活化、白细胞介素信号传导、Toll样受体级联反应和I-κB激酶/NF-κB信号传导等介导先天免疫系统活化和以及的过程。
这项研究通过精确定位技术,明确乳腺癌风险信号对应的基因突变,并定义了这些变异的靶基因。尽管大多数靶基因所对应的下游通路机制基础尚不清晰,但这对现有相关通路研究的方向作扩充和延伸,为乳腺癌易感性和发病机制等生物学研究奠定基础,并为临床作乳腺癌风险筛查提供强有力的证据!
参考文献:
Laura Fachal. Fine-mapping of 150 breast cancer risk regions identifies 191 likely target genes. Nature Genetics. 08 Jan, 2020.
Michailidou, K. et al. Association analysis identifies 65 new breast cancer risk loci. Nature, 2017.